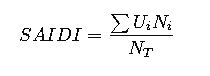Cálculo de inhibición enzimática (competitiva, no competitiva, acompetitiva) con precisión técnica
El cálculo de inhibición enzimática determina cómo los inhibidores afectan la velocidad de reacción. Este análisis es crucial para entender mecanismos enzimáticos y diseñar fármacos efectivos.
En este artículo, exploraremos fórmulas, variables y ejemplos prácticos para inhibición competitiva, no competitiva y acompetitiva. Además, se incluyen tablas con valores comunes y casos reales.
Calculadora con inteligencia artificial (IA) para Cálculo de inhibición enzimática (competitiva, no competitiva, acompetitiva)
- Calcular velocidad inicial (V0) con inhibidor competitivo a [S] = 0.5 mM, Ki = 0.1 mM, Km = 0.2 mM, Vmax = 100 µmol/min.
- Determinar Km aparente en inhibición no competitiva con Ki = 0.3 mM y [I] = 0.2 mM.
- Calcular V0 en inhibición acompetitiva con [S] = 1 mM, Ki = 0.05 mM, Km = 0.4 mM, Vmax = 150 µmol/min, [I] = 0.1 mM.
- Comparar efectos de inhibición competitiva y no competitiva con [S] = 0.8 mM, Km = 0.3 mM, Vmax = 120 µmol/min, [I] = 0.15 mM, Ki = 0.1 mM.
Tablas de valores comunes para Cálculo de inhibición enzimática
| Tipo de Inhibición | Variable | Valores Comunes | Unidad | Descripción |
|---|---|---|---|---|
| Competitiva | Km (constante de Michaelis) | 0.01 – 10 | mM | Concentración de sustrato para alcanzar Vmax/2 |
| Competitiva | Ki (constante de inhibición) | 0.001 – 1 | mM | Afinidad del inhibidor por la enzima |
| Competitiva | Vmax (velocidad máxima) | 10 – 500 | µmol/min | Velocidad máxima sin inhibidor |
| No competitiva | Km | 0.01 – 10 | mM | Constante de Michaelis sin cambio en inhibición pura |
| No competitiva | Ki | 0.001 – 1 | mM | Constante de inhibición para unión al complejo enzima-inhibidor |
| No competitiva | Vmax | 10 – 500 | µmol/min | Disminuye proporcionalmente con inhibidor |
| Acompetitiva | Km | 0.01 – 10 | mM | Disminuye en presencia de inhibidor |
| Acompetitiva | Ki | 0.001 – 1 | mM | Constante de inhibición para unión al complejo ES |
| Acompetitiva | Vmax | 10 – 500 | µmol/min | Disminuye proporcionalmente con inhibidor |
Fórmulas para Cálculo de inhibición enzimática y explicación detallada de variables
Inhibición competitiva
La inhibición competitiva ocurre cuando el inhibidor compite con el sustrato por el sitio activo de la enzima. La fórmula para la velocidad inicial (V0) es:
- V0: velocidad inicial de la reacción (µmol/min)
- Vmax: velocidad máxima sin inhibidor (µmol/min)
- [S]: concentración de sustrato (mM)
- Km: constante de Michaelis (mM), indica afinidad enzima-sustrato
- [I]: concentración de inhibidor (mM)
- Ki: constante de inhibición (mM), afinidad del inhibidor por la enzima
En inhibición competitiva, Km aparente aumenta debido a la competencia, mientras que Vmax permanece constante.
Inhibición no competitiva
En la inhibición no competitiva, el inhibidor se une a un sitio diferente al activo, afectando la función enzimática sin impedir la unión del sustrato. La fórmula es:
- V0: velocidad inicial (µmol/min)
- Vmax: velocidad máxima sin inhibidor (µmol/min)
- [S]: concentración de sustrato (mM)
- Km: constante de Michaelis (mM), permanece constante en inhibición pura
- [I]: concentración de inhibidor (mM)
- Ki: constante de inhibición (mM), afinidad del inhibidor por la enzima o complejo enzima-sustrato
En este tipo, Vmax disminuye proporcionalmente a la concentración de inhibidor, mientras que Km no cambia.
Inhibición acompetitiva
La inhibición acompetitiva ocurre cuando el inhibidor se une únicamente al complejo enzima-sustrato (ES), reduciendo tanto Vmax como Km. La fórmula es:
- V0: velocidad inicial (µmol/min)
- Vmax: velocidad máxima sin inhibidor (µmol/min)
- [S]: concentración de sustrato (mM)
- Km: constante de Michaelis (mM)
- [I]: concentración de inhibidor (mM)
- Ki: constante de inhibición (mM), afinidad del inhibidor por el complejo ES
Ambos parámetros, Km y Vmax, disminuyen en la misma proporción, manteniendo constante la relación Km/Vmax.
Variables comunes y su interpretación en inhibición enzimática
- Km (constante de Michaelis): Indica la afinidad de la enzima por el sustrato. Valores bajos indican alta afinidad.
- Vmax (velocidad máxima): La máxima velocidad alcanzable por la enzima cuando está saturada de sustrato.
- Ki (constante de inhibición): Mide la afinidad del inhibidor por la enzima o complejo ES. Valores bajos indican inhibidores potentes.
- [S] (concentración de sustrato): Variable experimental que afecta la velocidad de reacción.
- [I] (concentración de inhibidor): Concentración del inhibidor presente en la reacción.
Ejemplos prácticos de Cálculo de inhibición enzimática
Ejemplo 1: Inhibición competitiva en la inhibición de la acetilcolinesterasa
La acetilcolinesterasa es una enzima clave en la transmisión nerviosa. Supongamos que un inhibidor competitivo se añade para bloquear su actividad. Los parámetros son:
- Vmax = 200 µmol/min
- Km = 0.5 mM
- Ki = 0.1 mM
- [S] = 0.3 mM
- [I] = 0.2 mM
Aplicando la fórmula de inhibición competitiva:
La velocidad inicial disminuye significativamente debido a la competencia del inhibidor con el sustrato.
Ejemplo 2: Inhibición no competitiva en la deshidrogenasa láctica
Consideremos la deshidrogenasa láctica con los siguientes parámetros:
- Vmax = 150 µmol/min
- Km = 0.4 mM
- Ki = 0.2 mM
- [S] = 0.5 mM
- [I] = 0.1 mM
Usando la fórmula de inhibición no competitiva:
La velocidad máxima se reduce por la presencia del inhibidor, mientras que Km permanece constante.
Profundización en el análisis y aplicaciones avanzadas
El cálculo de inhibición enzimática no solo es fundamental para la bioquímica básica, sino que también es esencial en farmacología, toxicología y biotecnología. La correcta interpretación de los parámetros cinéticos permite:
- Diseñar inhibidores específicos para enzimas patógenas.
- Optimizar dosis de fármacos para maximizar eficacia y minimizar efectos secundarios.
- Entender mecanismos de resistencia en microorganismos y células cancerígenas.
- Desarrollar biosensores basados en actividad enzimática modulada.
Además, la modelación matemática y simulación computacional de inhibición enzimática permiten predecir comportamientos en sistemas complejos, facilitando la investigación y desarrollo en ciencias biomédicas.